前言
“蛋白拆分/重组系统”已经被应用在包括PPI分析和药物筛选、生物逻辑门、光遗传学、基因组编辑、蛋白质修饰和生物成像、诊断和治疗以及定向进化等多个领域。 图1. “蛋白拆分/重组系统”的原理和应用概述
图1. “蛋白拆分/重组系统”的原理和应用概述
蛋白拆分/重组系统是一种通过将蛋白质分割并重新组装来控制蛋白质和细胞活性的方法,分为条件激活系统和自发重组系统。在条件激活系统中,分裂的蛋白片段只在特定诱导因素(如光、化学物质或温度)存在时重新组装并变得活跃。这种系统能够在精确的时间和地点激活蛋白质,具有高精确度和可调节性,适用于基因编辑和治疗干预等应用。而自发重组系统则依赖蛋白片段的自组装能力,不需要外部触发器或重组模块,然而这种系统在控制蛋白活性方面的背景噪声较高,难以达到精确控制。代表性蛋白包括GFP和内含子,前者用于蛋白活性检测,后者用于有效的蛋白运输。
蛋白拆分/重组系统在这一领域表现出色,通过重组分开的功能蛋白片段,实现对PPI的动态分析。特别是在复杂膜蛋白网络和细菌细胞中,相应技术提供了精确的测量与实时可视化能力。在药物筛选方面,蛋白拆分/重组系统也展示了巨大的潜力,例如利用裂解型荧光素酶进行PPI检测及激酶抑制剂筛选,BACTH系统与内肽系统提供了新的检测途径。此外,整合的裂解型TurboID系统和三部分裂解型GFP系统引入了高通量筛选技术,进一步推动了PPI调控药物的开发。
生物逻辑门是在生物系统内创建可编程电路的工具,用于实现外部控制和精确调节基因表达。通过结合蛋白拆分/重组系统,可以设计出更复杂的多输入逻辑门。例如,利用分裂的T7RNAP与温度敏感的TlpA结合,可以通过温度变化调控基因表达。还有分裂的基于aaRS的逻辑门,可以通过非天然氨基酸作为输入源展示潜在用途。近年来,利用嵌合蛋白开发创新生物逻辑门的应用得到扩展。例如,将分裂嵌合蛋白与分裂的Cas9结合,用于构建NAND门或生物AND门,可实现复杂的翻译调控和精确的交叉标记。
光遗传学策略通过将分裂蛋白与光响应重组模块(如VVD和Magnet)结合,实现了精确的控制机制。例如,分裂T7RNAP与Magnet融合,可调控大肠杆菌中的基因表达。分裂dCas9与Magnet融合,允许基因靶点的可逆光学调控,促进神经元分化。进一步应用还表现在利用分裂的dCas12a进行受控基因组编辑,或者通过蓝光和红光激活系统实现实时基因敲除和病理生理机制的重组处理。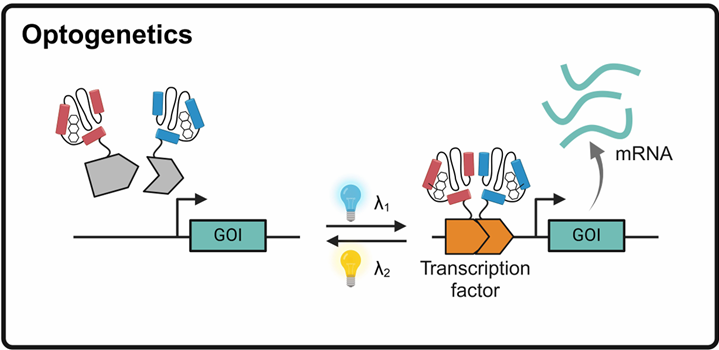
分裂基因编辑工具在可诱导基因编辑控制方面引起了广泛关注。例如,利用光遗传学控制分裂的Cas9和Cre重组酶,实现基因编辑的时空精确性。通过分裂Cas13b/dCas13b与光化学激活相结合,展示了新的RNA编辑平台。分裂系统的尺寸缩减优势被用于AAV载体递送基因编辑工具至小鼠视网膜,实现高效基因编辑。此外,创新策略还包括设计增强精度和特异性的分裂酶系统,如在雷帕霉素激活下的分裂胞嘧啶脱氨酶系统和高活性的分裂ADAR2系统。这些系统显示出较高的编辑精度和目标活性,同时减少脱靶效应。
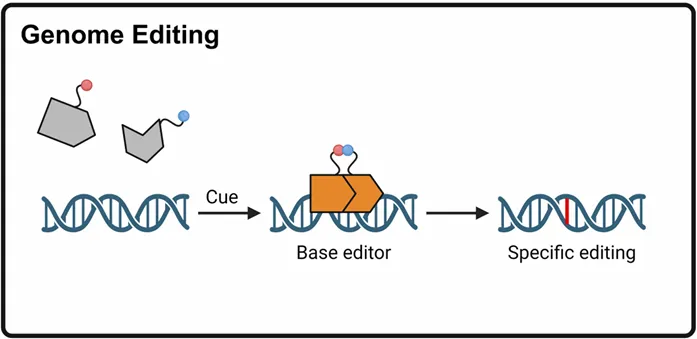
蛋白拆分/重组系统显著推进了蛋白质相互作用和细胞通讯的研究,最新研究展示了其应用。Martell等人利用分裂HRP标记与生物素相互作用的靶蛋白,有利于神经元突触的可视化,揭示了细胞间通讯机制。分裂-TurboID技术改进了该方法,适用于亚细胞结构或细胞边界蛋白质组学定位。分裂sfCherry和NanoBiT系统在膜界面和蛋白质聚集的疾病相关突变识别上发挥了作用。分裂β-内酰胺酶与抗体的整合,用于特异性分析物的酶检测和定量。串联蛋白反式剪接(tPTS)方法利用蛋白拆分/重组系统将修饰肽段整合到蛋白质中,突显了蛋白拆分/重组系统在蛋白质修饰和生物成像中的关键作用。

蛋白拆分/重组系统在疾病诊断和治疗相关的PPIs检测中发挥重要作用。基于内含肽介导分裂的分裂Cas9可检测癌症起源或细胞融合事件。在治疗方面,分裂β-内酰胺酶与癌症生物标志物融合,可在目标部位重新组装并激活药物。paCas9系统通过光遗传学精确破坏抗凋亡基因survivin,实现肿瘤体积缩减。分裂TEV蛋白酶系统控制了治疗性蛋白质的释放,如胰岛素和分泌工程稳定镇痛肽等。分裂dCas12a平台具有检测癌细胞的能力,凸显了蛋白拆分/重组系统在治疗策略中的重要地位。
分裂系统在定向进化研究中起着关键作用,特别是在噬菌体辅助连续进化(PACE)策略中。该策略通过将诱导的PPIs或蛋白质-DNA相互作用转化为PACE中的基因III或基因VI,这种方法结合分裂内源酶系统,用于改善可溶性表达的蛋白的进化。此外,应用非连续版PACE,结合分裂内源酶和分裂dSpCas9,产生了具有扩展原间隔邻近基序(PAMs)范围的SpCas9变体,展示了分裂系统在基因编辑工具扩展中的适应性。分裂T7RNAP系统的优化进一步促进了PPI分子的快速进化,强调了分裂系统对于开发高特异性分子功能的重要性,这些进展突显了分裂系统在定向进化中的重要影响。
参考资料:
[1] Bae J, Kim J, Choi J, Lee H, Koh M. Split Proteins and Reassembly Modules for Biological Applications. Chembiochem. 2024 May 17;25(10):e202400123. doi: 10.1002/cbic.202400123. Epub 2024 Apr 17. PMID: 38530024.
[2] H. K. Jiang, N. L. Ambrose, C. Z. Chung, Y. S. Wang, D. Soll, J. M. Tharp, Proc. Natl. Acad. Sci. USA 2023, 120, e2219758120;
[3] J. E. Habel, Methods Mol. Biol. 2021, 2261, 357–379.
[4] K. F. Cho, T. C. Branon, N. D. Udeshi, S. A. Myers, S. A. Carr, A. Y. Ting, Nat. Protoc. 2020, 15, 3971–3999.
[5] R. Ullah, M. A. Shah, S. Tufail, F. Ismat, M. Imran, M. Iqbal, O. Mirza, M. Rhaman, PLoS One 2016, 11, e0153436.



图1. “蛋白拆分/重组系统”的原理和应用概述
